Marek Bodzioch
Zakład Neurogenetyki, Katedra i Klinika Neurologii, Uniwersytet Jagielloński Collegium Medicum
W pierwszej części artykułu przedstawiłem w zarysie postępy badań genetycznych, dzięki którym poznaliśmy molekularne patomechanizmy nie tylko rzadkich zespołów wrodzonych, ale również wielu powszechnie występujących chorób. W drugiej części chciałbym nieco bliżej omówić najnowsze metody badawcze, które ten postęp umożliwiły i które w niedalekiej przyszłości mogą sprawić, że poznawanie własnych, osobistych genów, będzie równie łatwe i powszechne jak surfowanie po Internecie za pomocą smartfona.
Przez długie lata poważnym ograniczeniem badań genomu był brak technik molekularnych, umożliwiających odpowiednią efektywność analizy. Genom człowieka składa się z ponad 3 miliardów par zasad, a o skali trudności odczytania tak obszernej informacji genetycznej najlepiej świadczy fakt, że zadanie to zajęło w ramach Human Genome Project 13 lat, pochłaniając ogromne koszty i angażując liczne ośrodki naukowe w kilku krajach. Projekt ten realizowano jednak w całości za pomocą klasycznej metody sekwencjonowania (czyli odczytywania sekwencji informacji genetycznej) opracowanej przez Sangera. Metoda ta nadal stanowi „złoty standard” sekwencjonowania i jest obecnie powszechnie wykorzystywana w rutynowej diagnostyce genetycznej, ale cechuje się małą wydajnością. Idealnie nadaje się do analizy pojedynczych genów, jest jednak zbyt kosztowna i pracochłonna, aby można ją było efektywnie zastosować do badania kilkudziesięciu czy kilkuset genów, nie mówiąc już o całym genomie. W praktyce medycznej sekwencjonowanie sangerowskie jest bardzo przydatne do weryfikacji rozpoznania klinicznego ustalonego na podstawie charakterystycznych objawów. U dziecka z niedrożnością smółkową, nawracającymi zakażeniami dróg oddechowych i nadmiernym wydzielaniem jonów chlorkowych w pocie możemy wykonać analizę sekwencji genu CFTR, poszukując swoistych mutacji, które potwierdzą kliniczne rozpoznanie mukowiscydozy. Wykrycie swoistych mutacji, poza ostateczną weryfikacją rozpoznania, pozwala również określić rokowanie (niektóre mutacje powodują cięższe zaburzenia, inne – słabsze), a także potwierdzić stan nosicielstwa u rodziców, ustalić ryzyko powtórzenia się choroby u następnych dzieci i przeprowadzić w razie zainteresowania rodziców diagnostykę prenatalną w kolejnej ciąży.
W wielu przypadkach mamy jednak do czynienia z o wiele mniej swoistymi objawami niż te występujące w przebiegu mukowiscydozy. Do częstych powodów wizyty w poradni genetycznej należą m.in.: opóźnienie rozwoju psychoruchowego, autyzm, cechy dysmorfii (nietypowe cechy budowy twarzy lub całego ciała), ataksja móżdżkowa oraz przewlekłe polineuropatie lub dystrofie mięśniowe. Zaburzenia te mają bardzo liczne przyczyny genetyczne. Za pomocą klasycznych metod, takich jak sekwencjonowanie sangerowskie, jesteśmy w stanie wykluczyć tylko najczęstsze przyczyny genetyczne, np. u dzieci z niepełnosprawnością intelektualną i (lub) autyzmem – zespół Retta (u dziewczynek), zespół kruchego chromosomu X, zespół Angelmana i kilka innych zespołów, które łącznie odpowiadają za kilka, najwyżej kilkanaście procent tych przypadków.
Nadzieje na postęp diagnostyczny można wiązać z najnowszymi metodami biologii molekularnej, pozwalającymi realnie myśleć o niemal powszechnym dostępie do pełnej analizy osobistego genomu w ciągu najbliższych kilku lat. Już obecnie metody sekwencjonowania nowej generacji umożliwiają odczytanie pełnej sekwencji genomu jednej osoby w ciągu kilku tygodni za cenę poniżej 3000 USD. Czyli to, co w ramach Human Genome Project zajęto kilkanaście lat i kilka miliardów dolarów, dostępne jest teraz dla każdego, kogo stać na wydatek ok. trzech średnich krajowych pensji brutto. Jeżeli jest się naukowcem genetykiem i na dodatek kierownikiem dużego ośrodka naukowego w Stanach Zjednoczonych, sprawa jest jeszcze prostsza. W 2010 roku w „New England Journal of Medicine” ukazała się praca Jamesa R. Lupskiego i jego współpracowników, przedstawiająca wyniki sekwencjonowania całego genomu u jednego pacjenta z neuropatią dziedziczną. Tym pacjentem był sam Lupski, który, korzystając z dostępnych mu technologii, postanowił dowiedzieć się, jakie są przyczyny choroby nerwów obwodowych, występującej u niego i czworga jego rodzeństwa. We wcześniejszych badaniach wykluczył u siebie i swojego rodzeństwa najczęstsze przyczyny dziedzicznej polineuropatii. Dopiero sekwencjonowanie nowej generacji, tzw. sekwencjonowanie genomowe, pozwoliło mu znaleźć igłę w stogu siana, czyli jedną jedyną nieprawidłową parę zasad, spośród ponad 3 miliardów, w obrębie genu SH3TC2.
Podstawowym ograniczeniem w powszechnym zastosowaniu sekwencjonowania genomowego w diagnostyce genetycznej nie jest już raczej koszt, który zresztą szybko i systematycznie spada. Problem w tym, jak z gąszczu informacji genetycznej wyciągnąć tę właściwą, tę jedyną mutację, która odpowiada za chorobę jednogenową, jak u Lupskiego, czy być może zestaw jakichś szczególnych wariantów genetycznych determinujących podatność genetyczną na którąś z powszechnych chorób, takich jak miażdżyca, otyłość, udar czy choroba Alzheimera i inne postacie otępienia. Obecnie trwają intensywne prace nad sekwencjowaniem genomów coraz większej liczby ludzi. Z jednej strony sekwencjonuje się materiał genetyczny osób całkiem zdrowych, tak aby ustalić naturalną zmienność genetyczną, decydującą o normalnych różnicach między osobami, sprawiającą, że każdy z nas jest odrębną jednostką o swoich indywidualnych cechach wyglądu, charakteru i zdolności. Z drugiej strony bada się ludzi obarczonych różnymi chorobami, tymi jednogenowymi i wieloczynnikowymi, w poszukiwaniu unikatowych cech genetycznych, które byłyby odpowiedzialne za niekorzystne następstwa kliniczne. Ogromną zaletą tych badań jest możliwość odczytania pełnej informacji genetycznej, pozwalająca w zasadzie uniknąć sytuacji, kiedy wybiórczo skupiamy się na analizie jakiego fragmentu DNA, nieświadomi występowania innych ważnych zmian w genomie, które mogłyby być prawdziwą przyczyną obserwowanych u pacjenta zaburzeń.
Sekwencjonowanie genomowe dostarcza jednak olbrzymiej ilości danych, wymagających bardzo pracochłonnej analizy bioinformatycznej, tym bardziej skomplikowanej, że nie znamy jeszcze znaczenia zdecydowanej większości odczytywanej informacji genetycznej. Wciąż zatem podejmuje się próby racjonalnego ograniczenia zakresu analizy w celu jej uproszczenia i przyspieszenia. Popularne podejście polega na analizie eksomu zamiast całego genomu. Eksomem określa się wszystkie eksony kodujące białka. Sekwencja eksomu stanowi jedynie ok. 1,5% sekwencji całego genomu, na nią jednak, jak się uważa, przypada zdecydowana większości mutacji patogennych i wariantów polimorficznych o istotnym znaczeniu klinicznym.
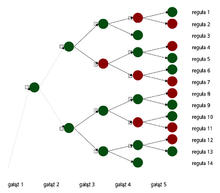
W badaniach podstaw chorób uwarunkowanych wieloczynnikowo nadal stosuje się metody analizy znanych i powszechnie występujących w populacji wariantów polimorficznych. Przykładem takiego badania był niedawno zakończony projekt pt. „Nowoczesna analiza swoistych wariantów genetycznych drobnocząsteczkowych modulatorów neurometabolizmu i neuroprotekcji w chorobach układu nerwowego”. W badaniu tym, zrealizowanym pod kierunkiem autora niniejszego artykułu, badaliśmy wpływ różnych polimorfizmów genetycznych w obrębie genów związanych z procesami przemian metabolicznych i cytoprotekcji w mózgu na ryzyko rozwoju chorób neurozwyrodnieniowych, takich jak choroba Parkinsona, udar niedokrwienny mózgu i padaczka. Porównywaliśmy częstość występowania około 250 wariantów polimorficznych u chorych oraz wśród odpowiednio dobranych pod względem płci i wieku zdrowych osób z grupy kontrolnej. Wyniki poddaliśmy analizie bioinformatycznej, której efektem było stworzenie tzw. drzewa decyzji, opisującego zestawy reguł różnicujących osoby zdrowe i chore. Reguły te wskazują, jakie układy badanych wariantów genetycznych wiążą się ze stanem choroby, a jakie ze stanem zdrowia. Schemat takiego drzewa decyzji przedstawia w uproszczeniu rycina 1., na której czerwone koła ilustrują reguły determinujące chorobę Parkinsona, zielone zaś odpowiadają regułom, które najwyraźniej opisują genetyczną odporność na zachorowanie na tę chorobę.
Doceniając niespotykane dotąd możliwości technik sekwencjonowania nowej generacji, należy wspomnieć i o tym, że mimo niewątpliwych zalet nie są one jednak wystarczające do uzyskania pełnej informacji o genomie badanej osoby. Nie chodzi nawet o to, iż pewnych regionów chromosomów, obejmujących głównie nieaktywną transkrypcyjnie heterochromatynę centromerów i telomerów, praktycznie nie udaje się sekwencjonować za pomocą dostępnych współcześnie technologii. Ważniejsze jest to, że istnieje jeszcze jeden bardzo istotny typ zmienności genetycznej, inny niż mutacje czy polimorfizmy dotyczące pojedynczych par zasad, polegający na zmiennej liczbie kopii różnych regionów genomu.
Większość genów występuje w dwóch kopiach, po jednym na każdym chromosomie z danej pary, ale mężczyźni mają akurat po jednej kopii genów umiejscowionych na chromosomach X i Y (poza tzw. regionem rzekomo autosomalnym, który jest reprezentowany dwiema kopiami u każdej płci – u mężczyzn druga kopia tego regionu znajduje się na chromosomie Y). Zróżnicowana liczba kopii genów w zależności od płci to najczęstsza, a na pewno najbardziej znana postać tego typu zmienności genetycznej. Genom zawiera jednak dużo więcej regionów cechujących się tego typu zmiennością. Badania w tej dziedzinie pozostają nieco w cieniu postępów sekwencjonowania, ale również tutaj nastąpiły w ostatnich latach przełomowe zmiany dzięki opracowaniu coraz doskonalszych metod mikromacierzowych.
Najczulszą metodą analizy liczby kopii genów w obrębie całego genomu jest analiza porównawczej hybrydyzacji genomowej z zastosowaniem mikromacierzy dużej gęstości, zawierających sondy genetyczne reprezentujące poszczególne fragmenty każdego z chromosomów. Ich więcej sond, tym większa czułość analizy. Najdokładniejsze dostępne obecnie mikromacierze zawierają jeden milion, a nawet więcej osobnych sond, pozwalają stwierdzić niewykrywalne za pomocą sekwencjonowania delecje i duplikacje o wielkości nawet kilkudziesięciu par zasad. Analiza polega na porównaniu DNA badanej osoby z DNA wzorcowym (np. DNA osoby zdrowej i chorej). Próbki DNA znakuje się barwnikami fluorescencyjnymi, np. chorego – barwnikiem zielonym, a zdrowego – czerwonym. Następnie obie próbki miesza się na płytce mikromacierzowej, gdzie poszczególne fragmenty DNA przyłączają się do odpowiednich sond. Wszędzie tam, gdzie regiony chromosomowe u chorego i zdrowego występują w równej liczbie kopii, na płytce mikromacierzowej pojawi się sygnał żółty (jako połączenie fluorescencji zielonej i czerwonej). Jeżeli jednak w danym regionie u pacjenta występuje delecja, proporcjonalnie mniejsza będzie ilość pochodzącego od niego DNA, reprezentującego ten fragment genomu. W tym miejscu pojawi się zatem względna przewaga fluorecencji czerwonej pochodzącej z DNA kontrolnego. Odwrotna sytuacja zdarzy się w przypadku występowania u pacjenta amplifikacji (czyli zwiększenia liczby kopii danego fragmentu genomu). Wówczas w miejscu amplifikacji przeważać będzie fluorescencja zielona pochodząca z DNA pacjenta.
U kilkuletniej dziewczynki przedstawionej na rycinie 2. przeprowadziliśmy analizę porównawczej hybrydyzacji genomowej za pomocą mikromacierzy zawierającej 135 tys. sond. Powodem wykonania badania były liczne cechy dysmorfii (płaska twarz, ubytek tęczówki w prawym oku i pojedyncza górna jedynka), opóźnienie rozwoju psychoruchowego, obustronne ubytki kości ciemieniowych (ryc. 3.) oraz napady padaczkowe. Rycina 4. pokazuje wyniki analizy dla chromosomu 13. Wykres przedstawia stosunek fluorescencji dwóch barwników, którymi wyznakowano DNA badanej dziewczynki i DNA kontrolne. W regionie 13q32.3-13q33.1 stwierdziliśmy znaczny nadmiar sygnału pochodzącego z DNA dziecka, wskazujący na zwielokrotnienie obszaru o długości około 4,6 milionów par zasad. Dokładniejsze analizy potwierdziły, że obszar ten występuje u dziewczynki aż w czterech kopiach. Obejmuje on kilkanaście genów o większości nieznanych dokładnie funkcjach. Na uwagę zasługuje obserwacja, że wśród nich znajduje się m.in. gen NALCN, kodujący kanał jonowy przepuszczający jony sodowe, podlegający ekspresji w mózgu. Dotychczas nie zidentyfikowano mutacji tego genu u ludzi, niemniej jednak jego zwielokrotnienie (cztery kopie zamiast dwóch) mogłoby tłumaczyć występujące u dziewczynki napady padaczkowe, które można by przypisać zwiększonej ekspresji tego genu i wynikających z niej zaburzeń czynności elektrycznej komórek kory mózgowej. W ten sposób wyniki nowoczesnych badań całego genomu u dziecka z rzadkim zespołem genetycznym przyniosły wskazówkę, sugerującą związek genu NALCN z patogenezą padaczki u ludzi. Dalsze badania pozwolą ustalić, czy mutacje lub warianty polimorficzne tego genu wiążą się ze zwiększonym ryzykiem napadów padaczkowych w ogólnej populacji.
Mam nadzieję, że ten krótki przegląd przybliżył nieco Czytelnikom najnowsze kierunki badań w genetyce. Dziedzina ta jest obecnie przedmiotem bardzo dynamicznego i wielokierunkowego rozwoju, który z pewnością będzie coraz silniej wpływał na współczesną medycynę, a nie można wykluczyć, że jego skutkiem będą również ważne następstwa społeczne.
Artykuł przygotowano z pomocą grantu N R13 0038 04 Ministerstwa Nauki i Szkolnictwa Wyższego.